【ツール】
今回使用するツールは、
- alvaDesc 2.0.12:分子記述子とフィンガープリント(FP)の計算
- alvaModel 2.0.4:alvaDescで計算された結果を使い回帰・分類モデルの作成
- alvaRunner 2.0.4:alvaModelで作成されたモデルの化合物データセットへの適用
の3つです。
【データセット】
データセットは、モデルの作成と検証に使うものと、作成したモデルに適用するものの2つを使用します。
1. モデル作成に使用するデータセット:dataset_KM_ARNOT.txt(ヘッダー付きSMILESファイル)
-
VEGA ToolkitのEcological PBPK(生物学的薬物速度論)セクションにある、kM/Half-Life
model (Arnot-EpiSuite)の回帰モデル用の421個のトレーニングデータと210個のテストデータを含む、計631個のデータセットです。エンドポイントkM/Half Lifeの実験値は魚類に於ける生体内変換率で、取り込んだ化学物質の半減期(日)の常用対数をとった速度定数kMになります。
-
このデータセットに含まれるテストデータ210個を、予めランダムに105個ずつに分け、一方をトレーニングデータに付加してモデル作成用の526個のデータセットとし、もう一方をモデル作成後の検証用としました。
-
このデータセットは、VEGAの無料JAVAアプリ”VEGA-GUI”をダウンロードすることで入手できます。
https://www.vegahub.eu/download/vega-qsar-download/
-
VEGAの使い方については、弊社ブログ「VEGA Toolkit:VEGA-GUI-1.2.0の使用方法」をご覧ください。
https://www.affinity-science.org/2022/03/vega-toolkit-vega-gui-1.html
2. 作成したモデルを適用するデータセット:Chemical_List_40CFR1164.txt(ヘッダー付きSMILESファイル)
-
アメリカ合衆国環境保護庁(EPA)より入手した米国の連邦水質汚染防止法に関連する有害物質“40 CFR 116.4 Designation of Hazardous Substances” 333化合物の内、SMILESレコードがある290化合物のデータセットです。
-
このデータセットはEPAのサイトの”Lists”から”Lists of Chemicals”を選択、表示されるリストの中から”40CFR1164” (有害物質 333化合物)を選択、画面右側のEXPORTからcsvを選択することでcsvファイルをダウンロードすることができます。
https://comptox.epa.gov/dashboard/
【alvaDescによる記述子・FP計算とプロジェクトファイル出力】
それでは、まずalvaDescに526分子のKM_ARNOTデータを入力して記述子とFPを計算させます。
入力データのフォーマットがSMILESのため3次元情報を必要とする記述子は計算できませんので、alvaDescが持つ5666個の記述子の内、4179個の記述子を計算しました。
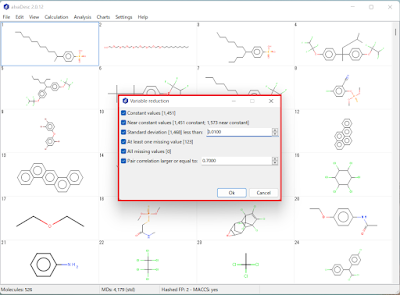
・変数削減ツールの活用
また、その4179個全部をalvaModelに投入すると計算効率が悪いので、記述子を保存する際に登場する変数削減ツールを活用します。[図1]
全ての分子について値が同じ記述子を削除、1つを除いて他の分子が同じ値のものを削除、標準偏差が0.01以下のものを削除、少なくとも欠損値が1つあるものを削除、全てが欠損値であるものを削除、ペア相関が0.7以上であるもの(の片方)を削除。
もう一度記述子の保存メニューを開くと、4179個の記述子が412個に減っていることがわかります。[図2]
・外部変数のインポート
この状態でalvaDescのプロジェクトファイルに保存しても良いのですが、ここでは、alvaDescの外部変数インポート機能を使って、目的変数などの情報を追加しておきます。[図3]
取り込まれた外部変数は、[View]>[Grid]からmolecule worksheetを開いて確認することができます。[図4]
外部変数の取り込みを行ったらalvaDescプロジェクトファイルに出力し、alvaDescのお仕事は終了です。
【alvaModelによるモデル作成】
・データの分割(スプリット)
alvaDescで保存したプロジェクトファイルをalvaModelにインポートし、まずデータをトレーニングとテストに区分けします。
alvaModelにデータがインポートされた状態ではMasterという1つの属性のデータセットとして扱われていますので、alvaDescに取り込んだ外部変数に入っているstatusというカラムのTraining/Test区分を使ってTrainingとTestというサブセットに分けます。[図5]
・モデルの手動生成
さて、いよいよ回帰モデルの作成に入る訳ですが、alvaModelの大きな特徴である多数の記述子を使ったモデルの自動生成を行う前に、FPを使用して手動でモデル生成を行ってみます。このステップは特に必要ではありませんが、alvaModelに実装されている4つの回帰モデルのどれを使うかのアタリをつけることもできるかと思ってやってみます。
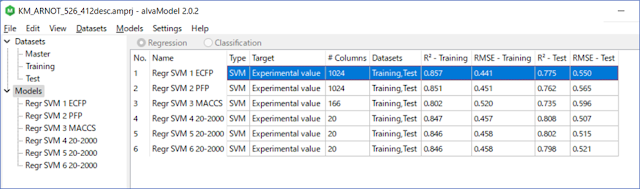
ここでは、ECFP/PFP/MACCS166の3種類のFPに対してSVMを使ってみました。その結果を見ると、モデルRegr SVM 1 ECFPがトレーニングのR2が0.857、RMSEが0.441と3つの中では一番良い結果となっています。[図6]
・モデルの自動生成
SVMはモデル作成手法として悪くはなさそうなので、SVMを使い自動生成をします。
Feature selectionのメニューで説明変数である記述子の最大値を20、ポピュレーションの大きさを120、繰返しを2000、最大化を図るスコアをR2に設定して自動生成を走らせてみます。[図7]
説明変数の数はいくら大きな数でも設定できますが、大きくすると過学習となってしまうこともありますので、いくつか大きさを変えて走らせてみた方が良いかと思います。
トレーニングに含まれる分子の数や設定した説明変数の数、モデルの種類などにより自動生成にかかる時間は変わります。最初に繰返し数を大きな値に設定しておき、進行具合のグラフ表示を随時見に行って、スコアの伸びが殆どなくなって来たらPauseボタンを押してストップさせることも有効な方法です。[図8]
今回は、設定した2000回の繰返しが終わった後でスコア順に並んだモデルの中から3つのモデルを保存しました。No.1とNo.2は殆ど同じようなので、No.1、No.3、No.4の3つにしました。[図9]
こうして生成された6つのモデルを比べてみると、20個の記述子を使ったRegr SVM 4 20-2000のモデルはトレーニングのR2が0.847、RMSEが0.457とRegr SVM 1 ECFPよりも悪いのですが、テストではその逆になっており、記述子を使ったSVMモデルも良い感じです。[図10]
・説明変数の数を変えたモデルの自動生成
次に、説明変数を15個に減らして自動生成を実行させてみます。
2000回の反復が終了した後で表示された結果を見て、No.1、No.3、No.5を保存することにしました。[図11]
このケースの場合、説明変数となる記述子の数を15個に減らしてもスコアの値は殆ど変わらないことがわかります。[図12] 同程度の性能であれば、説明変数の数がより少ない方がモデルとして効率が良いとも考えられます。
今回とは別のデータで、トレーニングとテストのスコアと説明変数の数の関係を見るために、説明変数の数を少しずつ変えてモデル生成を行ってみたことがあります。その結果をMS Excelを使用してトレーニングとテストのスコア(R2)をY軸、説明変数の数をX軸としたグラフにしました。青色で示したモデル毎のトレーニングのR2は説明変数の数を増すとどんどん大きくなりますが、同時に橙色で示したテストのR2が大きく減少していくことがわかります。ちなみに、グレー色で示したトレーニングとテストの平均を見ると、この場合、説明変数20個あたりが準最適と言っても良いのかもしれません。[図13]
このように、時間が許せば目的変数の数や選択するモデルの種類などを変えながら自動生成を行い、準最適パラメータを探索することも有効と思われます。
・自動生成で採用された記述子の比較
ここで、記述子を使って生成されたモデル4・5・7・8について、モデル毎に採用された記述子がどの程度に似通っているかを見てみたいと思います。alvaModelのモデル情報画面で右クリックをするとモデル情報がコピーできるので、それをエクセルに張り付けて比較してみます。[図14]
この4つのモデルでは、トレーニングでのR2とRMSEの値は殆ど変わりませんが、使われている記述子は違っていることがわかります。モデル4と5は使われている記述子20個の内13個が同じものでした。また、モデル7と8では、使われている記述子15個の内11個が同じものでした。これはalvaModelでの自動生成では記述子選択を遺伝的アルゴリズム(GA)による繰返しで行っていくため、性能の良いモデルが勝ち残っていくにつれ、互いに似通った記述子のセットが生き残っていくことが伺われます。
しかし、記述子20個のモデル4と15個のモデル7を比較すると、共通する記述子は2つしかありませんでした。但し、既にalvaDescの変数削減で相関の強い記述子を削減しているため、似た傾向を持つ別の記述子に置き換わっている可能性は低いと思われます。
記述子20個のモデルと15個のモデルは、別のGAプロセスで生成されましたので似ていないことは理解できますが、使用する記述子の数と種類が違っていても同程度の性能を持つモデルが生成されていることは留意すべきポイントかとも思われます。
・他の手法によるモデルの生成
他の手法でどのようになるかも試してみます。PLSとKNNでECFPを使って手動モデル作成を行うと、PLSはSVMに近いR2の値となりましたが、KNNでは低い値となりました。ちなみに、KNN法ではJaccard/Tanimotoの距離を使用しています。[図15]
記述子数を15にしてPLSとKNNで自動作成を走らせてみます。PLSではFPで作ったモデルより記述子で生成したモデルの方がスコアが低くなりましたが、KNNでは良くなっています。[図16]
後編ではモデルの処理、出来上がったモデルの検証と適用について説明していきます。
(後編に続く)